發布人:Google Health Genomics 產品負責人 Andrew Carroll 和 Genomics 項目經理 Howard Yang
Google Health 的 Genomics 團隊很高興分享我們對 DeepVariant 的最新擴展 - DeepTrio。
DeepVariant 于 2017 年首次發布,是一種開源工具,支持研究人員和臨床醫生分析個人的基因組測序數據并識別基因變異,如可導致疾病的變異片段。我們在 DeepVariant 方面的持續研究因其一流的準確性而獲得肯定。借助 DeepTrio,我們擴展了 DeepVariant,從而能夠在分析母親-父親-子女三人的基因序列數據時考量基因變異。
人類是二倍體生物,攜帶人類基因組的兩個副本。每個個體都分別繼承了來自母親和父親的一個基因組副本。父母遺傳信息有助于分析符合孟德爾遺傳規律的特征和疾病。DeepTrio 學習直接在測序數據中使用孟德爾遺傳的特性,以便在可以共同分析父母和子女樣本的情況下更準確地識別基因變異。
修改 DeepVariant 以分析三人樣本
DeepVariant 學習到如何將基因組中的位置分類為參考或者“基因組瀏覽器”(相關專家用于分析的工具)中數據表示的變種。“《使用 DeepVariant1.0 提高基因組分析的準確性》(Improving the Accuracy of Genomic Analysis with DeepVariant 1.0)”一文很好地概述了有關內容。
使用 DeepVariant1.0 提高基因組分析的準確性
https://ai.googleblog.com/2020/09/improving-accuracy-of-genomic-analysis.html
DeepVariant 接收數據作為以候選變異為中心的基因組窗口,并且按照要求將數據分類為參考(無變異)、雜合子(變異的一個副本)或純合子(兩個副本均為變異)。DeepVariant 將序列證據視為代表數據特征的通道(請參閱“《透過 DeepVariant 深入觀察》(Looking through DeepVariant’s eyes)”獲取更深入解釋)。
透過 DeepVariant 深入觀察
我們修改了 DeepTrio,以在單個圖像中表示來自三人的序列數據,其中每個樣本的高度固定,子女的數據置于中間。我們使用來自美國國家標準與技術研究院 (NIST) 瓶中基因組聯盟 (GIAB) 的黃金標準樣本作為真實標簽,訓練一個模型調用子女樣本中的變異,并訓練另一個模型調用頂部父母樣本中的變異。為調用父母雙方的樣本,我們翻轉父母樣本的位置。
衡量 DeepTrio 提高的準確性
我們研究發現,對于父母和子女變異檢測,DeepTrio 比 DeepVariant 更準確,在覆蓋率較低的情況下具有特別明顯的優勢。這使研究人員能夠以更高的準確性分析樣本,或者在顯著降低費用的同時保持相當的準確性。
為評估 DeepTrio 的準確性,我們借助 NIST 瓶中基因組聯盟提供的涵蓋廣泛特征的黃金標準,將其與 DeepVariant 的準確性進行比較。為獲得在訓練中從未見過的評估數據集,我們在訓練中排除了 20 號染色體,并對 20 號染色體進行了評估。
我們訓練 DeepVariant 和 DeepTrio,以對來自兩種不同儀器 Illumina 和 Pacific Biosciences (PacBio) 的數據進行測序。如要詳細了解這些技術之間的差異,請參見我們之前的博客。這些測序儀均以容易錯誤的方式隨機采樣基因組。為準確分析基因組,我們需要重復采樣相同區域。在某個位置的采樣深度稱為覆蓋范圍。以近似線性的方式測序來覆蓋更大的范圍將導致成本更高。因此我們經常需要在成本、準確性和測序樣本之間進行權衡。而權衡的結果是,在三人樣本中,父母樣本通常以較低的深度進行測序。
在下方圖表中,我們繪制了一系列不同覆蓋范圍中 DeepTrio 和 DeepVariant 的準確性。
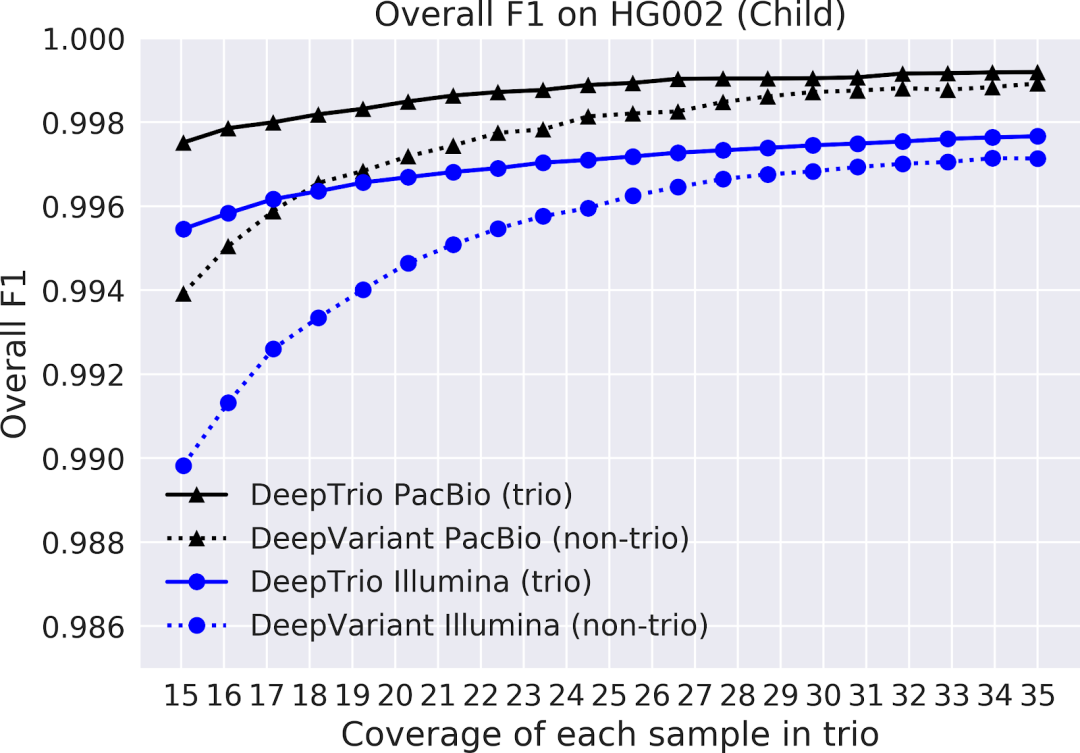
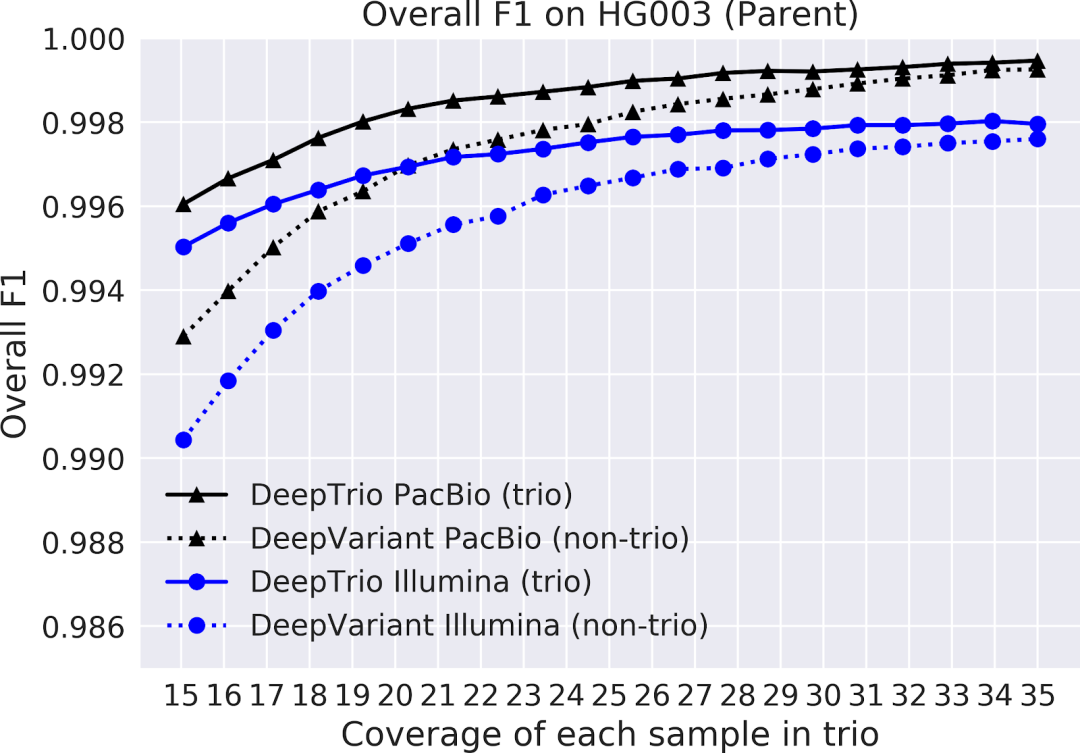
圖 2.子女樣本(頂部)和父母樣本(底部)上 DeepTrio(實線)和 DeepVariant(虛線)的 F1 得分,使用 Illumina(藍色)和 PacBio(黑色)儀器進行測序。在具有一系列測序覆蓋范圍(x 軸)的樣本中,對 20 號染色體上所有類型的小變異進行 F1 測量
DeepTrio 在 de novo 變異上的表現
相對于人類參考基因組,每個人大約有 500 萬處變異,其中絕大多數從父母處繼承而來。一小部分(大約 100 處)是新變異(稱為 de novo),由 DNA 復制過程中的復制錯誤導致。我們的研究證明,DeepTrio 大大減少了 de novo 變異檢測的假陽性情況。對于 Illumina 數據,其真陽性回收率的下降幅度較小,而對于 PacBio 數據,這種權衡不會發生。
為評估準確性,我們分析了父母兩人都被稱為非變異但子女被稱為雜合變異的位點。我們觀察到 DeepTrio 更不愿意將變異命名為 de novo,這類似于人類對于違反孟德爾遺傳規律的位點,需要獲得更高水平的證據。因此,此類 de novo 變異的假陽性率低得多,但 DeepTrio Illumina 的召回率則略低。通常,在發生這種情況時,子女的樣本仍將稱作變異,但父母樣本則給予“不調用”的判斷(分類器沒有足夠的信心進行調用)。
促進罕見病研究
通過將 DeepTrio 作為開源軟件發布,我們希望這一軟件能夠支持科學家更準確地分析樣本,進而提升對基因組數據的分析能力。我們希望這能促進研究和臨床開發,進而更好地解析罕見病病例,并改進治療方法的開發。
除了將 DeepTrio 的代碼作為開放源代碼發布之外,我們還發布了為訓練這些模型而生成的測序數據。這些數據會在預印本“《用于基準測試和開發的黃金標準樣本的廣泛序列數據集》(An Extensive Sequence Dataset of Gold-Standard Samples for Benchmarking and Development)”中加以說明。通過發布該生產模型和訓練相似復雜度模型所需的數據,我們希望能為基因組學界的方法開發做出貢獻。
原文標題:應用深度學習分析家庭基因組數據
文章出處:【微信公眾號:TensorFlow】歡迎添加關注!文章轉載請注明出處。
責任編輯:haq
-
開源
+關注
關注
3文章
3363瀏覽量
42543 -
深度學習
+關注
關注
73文章
5504瀏覽量
121239
原文標題:應用深度學習分析家庭基因組數據
文章出處:【微信號:tensorflowers,微信公眾號:Tensorflowers】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
如何提高OTDR測試的準確性
如何提高電位測量準確性
如何提升ASR模型的準確性
GPU深度學習應用案例
如何評估 ChatGPT 輸出內容的準確性
如何保證測長機測量的準確性?

AI大模型與深度學習的關系
【飛凌嵌入式OK3576-C開發板體驗】RKNPU圖像識別測試
HPLC數據怎么分析處理
影響電源紋波測試準確性的因素
深度學習模型訓練過程詳解
8芯M16插頭數據傳輸的準確性

測量時鐘信號的探頭要求:確保準確性與穩定性
電流探頭測試小技巧:提高準確性和安全性





 應用深度學習分析提高基因組分析的準確性
應用深度學習分析提高基因組分析的準確性











評論